SARS-CoV-2 steht in allen Schlagzeilen. Millionen von Diagnosekits und -reagenzien müssen an jeden Winkel des Planeten gelangen. Die wenigen Firmen können der weltweiten Nachfrage kaum noch nachkommen. Die Wartezeit für diese unerlässlichen Diagnosekits beträgt mehrere Wochen. Hinter den Kulissen der verlassenen Straßen, verborgenen Gesichtern und verflüchtigten Emissionen arbeitet ein Innsbrucker Laborteam fast unbemerkt an der Entwicklung eines alternativen PCR-Testverfahrens zur Diagnose des COVID-19 Erregers, mit dem die Ressourcenverknappung umgangen werden kann.
Von Umwelt-DNA zu Humanpathogenen

Eine “Multi-Channel”-Pipette erleichtert die Arbeit im Labor ungemein
Noch vor zwei Monaten beschäftigte sich Sinsoma GmbH, ein Spin-Off Unternehmen der Universität Innsbruck, vorrangig mit der DNA-Analyse im Umweltmanagement. Dies änderte sich schlagartig. Im Zuge der rapiden Ausbreitung von SARS-CoV-2 kam es zu einer umgehend benötigten Steigerung der Testkapazitäten. Das Team machte es sich demnach zum Ziel, eigene Protokolle zur Umwelt-DNA-Analyse abzuwandeln und diese an die Detektion der spezifischen viralen RNA in Humanproben anzupassen. Die Methode befindet sich derzeit in der finalen Validierungsphase und alle bisherigen Arbeiten verliefen äußerst erfolgversprechend.
Bei dieser Testmöglichkeit handelt es sich um eine Alternative zur Real-Time-PCR Methode, welche derzeit zur Diagnose des COVID-19 Erregers genutzt wird. Beide Testmöglichkeiten basieren auf demselben Prinzip der PCR.
„PCR“ – Was ist das überhaupt?
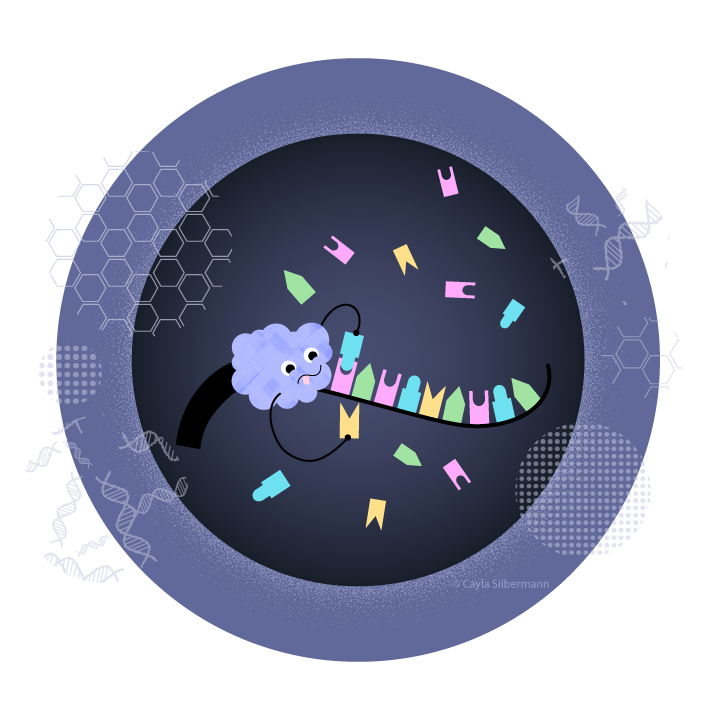
Die Abkürzung steht für Polymerase-Ketten-Reaktion (polymerase-chain-reaction). Seit ihrer bahnbrechenden Entwicklung von Kary Mullis im Jahre 1983 ist diese Methode eine nicht mehr wegzudenkende Retterin für jedes Berufsfeld, welches auch nur im Entferntesten mit DNA in Berührung kommt. Diese simple, jedoch raffinierte Reaktion erlaubt es DNA-Stücken, die zu bestimmten Arten gehören, millionen-, gar milliardenfach zu vervielfältigen, selbst, wenn in einer Probe nur wenige Zielmoleküle unter Millionen oder Milliarden anderen DNA-Molekülen vorhanden sind.
Das Prinzip ist einfach. Die PCR nimmt sich ein Beispiel an einem Prozess, der in jeder lebenden Zelle während der normalen Zellteilung stattfindet: Zuerst muss die doppelsträngige DNA in Einzelstränge aufgetrennt werden. Das Ziel ist es, zu jedem Einzelstrang eine ergänzende Kopie zu erstellen.
Die Stars der PCR-Show, die ihr auch den Namen verleihen, sind die sogenannten Polymerasen. Diese Enzyme nehmen sich einen Strang als Vorlage, docken dort an und rattern wie ein Zug auf Schienen entlang des Strangs. Gleichzeitig „kleben“ sie in Sekundenbruchteilen passende Bausteine wie ein Puzzle an den Strang an, sodass ein neuer Doppelstrang entsteht.
Es gibt aber ein Problem – die Polymerase weiß nicht, wo sie überhaupt anfangen soll. Sie benötigt ein Signal, welches sie an die richtige Stelle lotst. Hier kommen Primer ins Spiel. Primer sind kurze Einzelstränge, die speziell so entworfen wurden, dass sie nur auf jene Baustein-Sequenz der DNA passen, die man auch untersuchen möchte, beispielsweise auf die cDNA von SARS-CoV-2.
Wenn ein Primer sich an die DNA gebunden hat, bietet dieser der sonst verlorenen Polymerase eine Andockstelle, und zeigt ihr, wo sie anfangen soll, den neuen Strang herzustellen. Einen kompletten Ablauf dieser Schritte nennt man Zyklus. Um die gewünschte DNA millionenfach zu vervielfältigen, sind mehrere solcher Zyklen nötig.
Im diagnostischen Setting nutzt man Abwandlungen der PCR, um beispielsweise festzustellen, ob in einer Probe DNA oder RNA von Pathogenen zu finden sind. Wenn keine Infektion besteht, kann auch nach mehreren Zyklen kein Produkt entstehen, da die Primer die Bausteinabfolge des Erregers, für den sie spezifiziert wurden, nicht finden können. Ohne Primer gibt es keine Vervielfältigung durch die Polymerase. Ohne Vervielfältigung gibt es keine PCR-Produkte. Solche Testmöglichkeiten werden als vorgefertigte Testkits am Markt angeboten. Dies kann man sich wie einen fertigen „Chemiebaukasten“ vorstellen, bei dem die benötigten Materialien auch mitgeliefert werden. Im Labor muss dann nur noch die mitgelieferte Anleitung befolgt werden.
Wer genau in den Medien aufgepasst hat, ist vielleicht draufgekommen, dass es sich beim COVID-19 Erreger um einen RNA-Virus handelt. Um eine PCR zu ermöglichen, muss die RNA des Virus erst in DNA umgewandelt werden, was über ein besonderes Enzym, der Reversen Transkriptase, bewerkstelligt wird. Solche RNA, die umgewandelt wurde, wird als cDNA bezeichnet.
Same, same but different
Nun sind wir mit den PCR-Basics vertraut genug, um beide Verfahren ein wenig genauer unter die Lupe zu nehmen. Bei der qPCR, die derzeit für die Diagnose des COVID-19 Erregers genutzt wird, benötigt man zusätzlich zu den Primern, der Polymerase und den Bausteinen noch sogenannte „TaqMan-Sonden“. Fällt euch da was auf? Tatsächlich basiert die Namengebung der Sonde, die in den 90ern entwickelt wurde, auf dem Spiel „Pac-Man“. So erklärten die Entwickler die Interaktion der Polymerase mit der Sonde.
In diesem Beispiel handelt es sich um kurze Fragmente, die, wie die Primer auch, auf die cDNA des Virus abgestimmt sind. Sie „setzt“ sich auf genau den Bereich, den die Polymerase entlangrattern soll. Diese Sonde ist fluoreszent markiert, das bedeutet, dass sie kurz farbig aufleuchtet, wenn sie von einer Lichtquelle angeregt wird. Vorerst ist die Sonde noch inaktiv, da sie von einem anderen Molekül „beschattet“ wird. Erst, wenn die Polymerase wie „Pac-Man“ auf der DNA entlangwandert und die Sonde vom Strang trennt, wird die „Farbe“ freigesetzt. Wozu ist das gut? So kann die Menge der entstehenden cDNA von den qPCR Maschinen gemessen werden, während die Zyklen ablaufen. Deshalb heißt die Methode real-time PCR.
Je mehr cDNA in der Probe enthalten ist, desto mehr Licht wird freigesetzt. Ist die cDNA des COVID-19 Erregers nicht in der Probe, so können die Primer und Sonden nicht andocken. Die Polymerase kann somit nicht die Sonde von der DNA trennen und die Sonde emittiert kein Licht, da sie weiterhin „beschattet“ wird. In diesem Falle ist der Test also negativ.
Bei der von Sinsoma GmbH entwickelten Methode handelt es sich um ein Endpunkt-PCR-Verfahren mit anschließender Kapillarelektrophorese (CEL-PCR). Klingt kompliziert, ist es aber nicht! Es handelt sich hierbei um nichts weiter als um eine „klassische“ PCR, der noch ein Prozess angehängt wird, um das entstandene Produkt sichtbar zu machen. In diesem Falle wird die cDNA nach der PCR durch einen hauchdünnen „Schlauch“ (= Kapillare) geschleust. Dort kann mithilfe eines Lasers die mit einem Fluoreszenzfarbstoff versehene DNA gemessen werden, um zu sehen, ob ein Produkt entstanden ist.
Moment! Fluoreszenz – Hatten wir das nicht schon? Gut beobachtet. Der Unterschied ist, dass bei dieser Methode die Markierung in das PCR-Produkt eingebaut und nicht vom Einzelstrang abgespalten wird. Die CEL-PCR ist somit unabhängig von der Verfügbarkeit der qPCR Maschinen und den vorgefertigten Testkits, mit denen weltweit der Großteil der Testungen auf SARS-CoV-2 durchgeführt werden. So könnte das Innsbrucker Labor mit dieser Methode mehr als 1000 zusätzliche Testungen pro Tag durchführen.
Laut Professor Michael Traugott – Professor an der Universität Innsbruck und Mitbegründer der Sinsoma GmbH – kann das CEL-PCR Verfahren von Unternehmen verwendet werden, die ihre Mitarbeiter testen wollen. Professor Traugott meinte diesbezüglich: „Wir sind hier auch im Austausch mit Initiativen, die sich gebildet haben, und dem Gesundheitsministerium. Derzeit wird besprochen, wie in Österreich weitergemacht werden soll und wo in Österreich größere Screening-Testkapazitäten benötigt werden.“
Forschung steht in dieser Zeit still
Die derzeitige Krise wirkt sich stark auf jene Forschung aus, die nichts mit SARS-CoV-2 zu tun hat. Diese war bisher aufgrund der regulatorischen Maßnahmen nur auf Heimarbeit beschränkt. Feld- und Laborarbeiten waren bisher somit nicht möglich. Dabei ist es nicht jeder Forschungsgruppe möglich, sich der SARS-CoV-2 Pandemie anzupassen, da Projekte schon im Vorhinein von Forschungsgeldern bestimmt werden und somit ein „Ablaufdatum“ haben. Viele wissenschaftliche Bereiche, die vor der Krise noch im Gange waren, stehen deshalb derzeit still. Vor allem sind Forscherinnen und Forscher betroffen, deren Verträge befristet sind.
Ähnlich ergeht es der Forschungsgruppe, die von Michael Traugott neben der Mitarbeit in Sinsoma geleitet wird: „Die WissenschaftlerInnen bekommen natürlich im Projekt ihr Gehalt gezahlt. Sie können nicht in Kurzarbeit geschickt werden wie das bei meinen MitarbeiterInnen bei Sinsoma der Fall ist, sondern diese Mittel sind dann einfach weg“, erklärt Professor Traugott. Seiner Forschungsgruppe wurde vor der Krise ein Projekt genehmigt, um Nahrungsnetze in Agrarökosystemen zu erforschen. Dies solle eine Grundlage bieten, mögliche Wege zu finden, um in der Landwirtschaft Schädlingsregulation statt Pestiziden mit natürlichen Gegenspielern zu betreiben. Dies bedarf allerdings Forschungsarbeiten im Freiland, die derzeit nicht beziehungsweise nur eingeschränkt mit besonderen Auflagen erlaubt sind.
Michael Traugott vergleicht die derzeitige Situation mit einer Bergtour. „Es gibt Phasen im Leben, in denen es wichtige Aufgaben gibt, Chancen gibt, an denen man dranbleiben muss. Es ist vergleichbar mit einer Bergtour, die man zum ersten Mal geht und nicht genau weiß, wie der Weg aussehen wird. Es kann auch ein Vorteil sein, nicht genau zu wissen, was einen erwartet, denn würde man es wissen, hätte man eventuell zu viel Sorge, abzustürzen. Bei jeder Tour gibt es auch steile Phasen. Da muss man auch durchhalten und man geht an sein Limit, und bekommt dadurch die Chance, seine Fähigkeiten zu erweitern. In anderen Phasen wird es wieder flacher und da sollte man schauen, dass man sich wieder regeneriert.“

Das Team hinter Sinsoma, von links nach rechts: Dominik Kirschner, Christiane Zeisler, Martina Nindl, Michael Traugott, Corinna Wallinger, Daniela Sint. Nicht abgebildet: Karin Wastian